|
[研究概要]
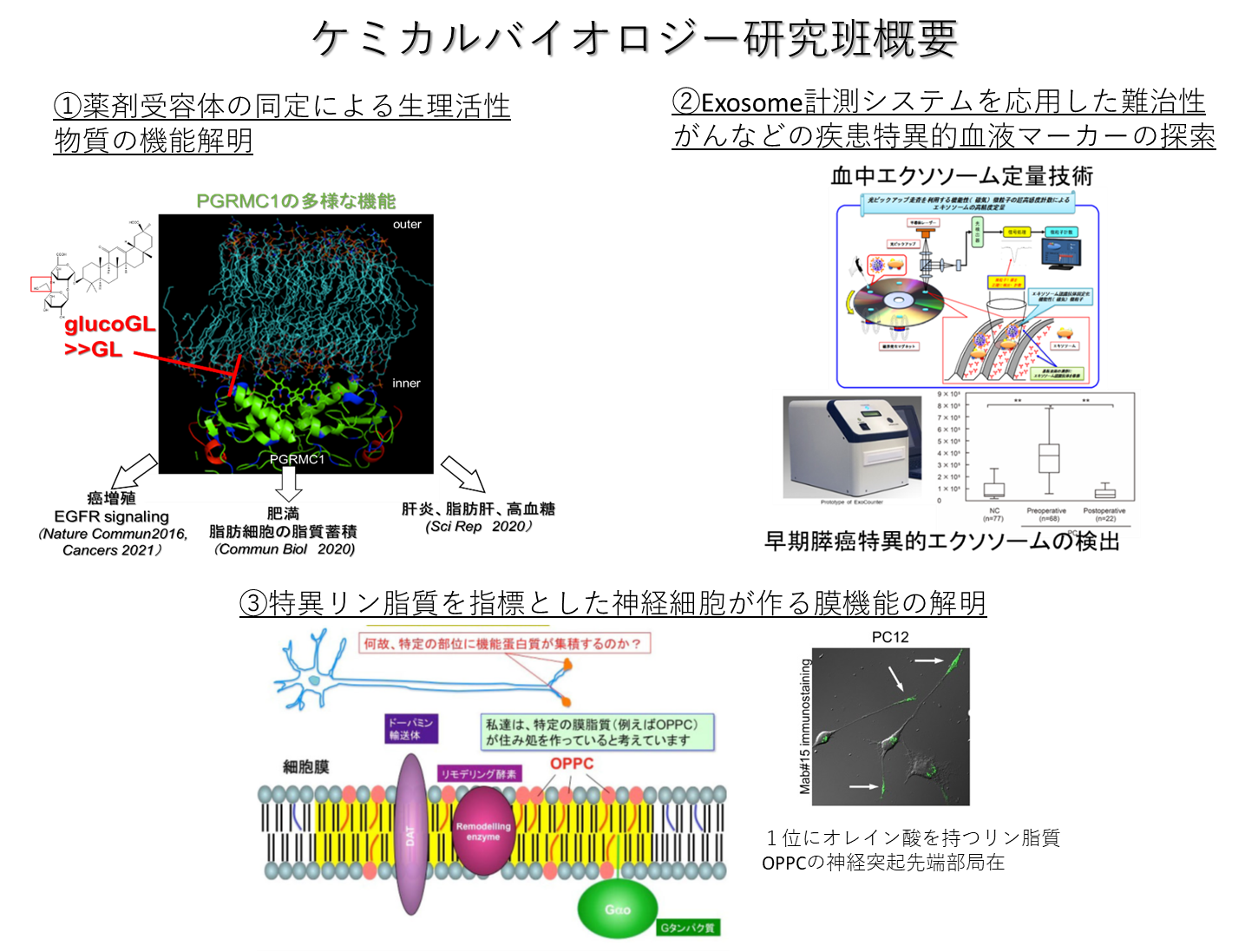
本研究班では、ナノビーズテクノロジーを利用した薬剤の標的となる受容体タンパク質の精製技術とこの受容体情報を指標としたケミカルバイオロジー解析、さらにナノビーズ技術と光センサーデバイスを融合した独自のバイオセンサーExosome計測技術を臨床応用した疾患マーカーの解析を行っています。また、脂質生化学的な手法を用いて細胞膜機能を制御するメカニズムの解明や、細胞膜上の構成分子の同定とその動態解析、細胞膜由来の細胞外分泌小胞の定量解析は、細胞膜を介する細胞機能全般やその異常などの未知の生理機能の解明や病態における関わり解析しています。これらの解析を通して、基礎医学、創薬、臨床展開を見据えた研究を進めて行きます。
具体的な研究内容としては、以下の大きく三つ研究テーマを遂行しています。
①天然有効化合物の受容体タンパク質の探索と、候補受容体PGRMC1の機能制御の解析を通じた癌増殖、肝機能制御、炎症応答の解明
②細胞外分泌膜小胞エクソソームの絶対定量システムを応用した疾患特異的血液マーカーの探索
③特異リン脂質OPPCを指標とした「局所的リン脂質脂肪酸リモデリング」を介した神経細胞が作る膜機能解明
|

